DNA - CS50x 2023
编写一个程序,根据DNA信息来识别个体身份。
$ python dna.py databases/large.csv sequences/5.txt
Lavender
开始
请登录 cs50.dev,点击终端窗口,然后输入 cd 命令。您会看到终端提示符如下:
接下来执行
wget https://cdn.cs50.net/2022/fall/psets/6/dna.zip
以便将 dna.zip 下载到您的 codespace。
然后执行
来创建名为 dna 的文件夹。创建完成后,您可以执行
并在提示符后输入 y,然后按回车键删除该 ZIP 文件。
现在输入
然后按回车键进入该目录。您的提示符现在应类似于以下内容。
单独执行 ls,您应该会看到一些文件和文件夹:
databases/ dna.py sequences/
如果遇到问题,请重复以上步骤,检查是否有遗漏或错误!
背景
DNA,作为生物遗传信息的载体,在刑事司法领域已经应用了几十年。那么,DNA分析是如何工作的呢?给定一段DNA序列,法医人员如何确定其身份?
实际上,DNA是由称为核苷酸的分子按特定顺序排列形成的(双螺旋结构)。每个人的细胞都有数十亿个按顺序排列的核苷酸。DNA 的每个核苷酸包含四种不同的碱基之一:腺嘌呤 (A)、胞嘧啶 (C)、鸟嘌呤 (G) 或胸腺嘧啶 (T)。DNA序列中,部分(即基因组)在人类中几乎相同或非常相似,但其他部分具有更高的遗传多样性,因此个体差异较大。
短串联重复序列 (STR) 是 DNA 中遗传多样性较高的区域。STR 是一段短的 DNA 碱基序列,在人体 DNA 的特定位置会连续重复多次。不同个体之间,特定 STR 的重复次数差异很大。例如,在下面的 DNA 样本中,Alice 的 DNA 中 STR AGAT 重复了四次,而 Bob 的 DNA 中相同的 STR 重复了五次。
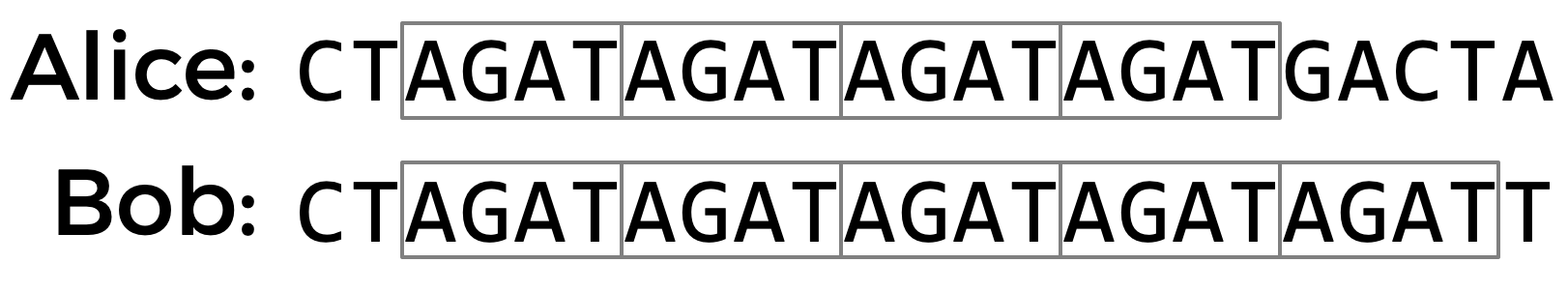
使用多个 STR 而不是仅使用一个 STR 可以提高 DNA 分析的准确性。如果两个人单个 STR 重复次数相同的概率为 5%,分析师检测 10 个不同的 STR,那么两个 DNA 样本偶然匹配的概率约为 1/1 千万亿(假设各 STR 之间相互独立)。因此,如果两个 DNA 样本在每个 STR 的重复次数上匹配,分析师可以非常有信心地认为它们来自同一个人。CODIS,即联邦调查局 (FBI) 的 DNA 数据库,使用 20 种不同的 STR 进行 DNA 分析。
这样的 DNA 数据库可能是什么样的?嗯,以最简单的形式,您可以想象将 DNA 数据库格式化为 CSV 文件,其中每一行对应于一个个体,每一列对应于一个特定的 STR。
name,AGAT,AATG,TATC
Alice,28,42,14
Bob,17,22,19
Charlie,36,18,25
上述文件数据表明,Alice 的 DNA 中,AGAT 连续重复 28 次,AATG 重复 42 次,TATC 重复 14 次。类似地,Bob 的这三个 STR 分别重复 17、22 和 19 次,Charlie 则分别为 36、18 和 25 次。
那么,拿到一段DNA序列后,如何确定它属于谁呢?假设你在DNA序列中找到了AGAT的最长连续重复序列,发现它重复了17次。如果再发现AATG的最长重复序列是22次,TATC的最长重复序列是19次,那么就很有可能这段DNA属于Bob。当然,也可能计算出的STR重复次数与DNA数据库中的任何人都无法匹配,这时就找不到匹配结果。
实际上,因为分析人员知道STR位于哪个染色体和DNA的哪个位置,所以他们可以将搜索范围缩小到DNA的一小段区域。不过,为了简化这个问题,我们先忽略这个细节。
你的任务是编写一个程序,输入一段DNA序列和一个包含个体STR计数的CSV文件,程序会输出这段DNA最有可能属于谁。
规范
在一个名为dna.py的文件中,实现一个程序,该程序可以识别DNA序列属于谁。
- 程序需要你提供两个命令行参数:第一个是包含个体STR计数的CSV文件名,第二个是要识别的DNA序列的文本文件名。
- 如果程序运行时提供的命令行参数数量不正确,程序应该打印一条错误信息(用
print语句)。如果提供了正确数量的参数,你可以假定第一个参数确实是有效CSV文件的文件名,第二个参数是有效文本文件的文件名。
- 如果程序运行时提供的命令行参数数量不正确,程序应该打印一条错误信息(用
- 你的程序应打开CSV文件并将其内容读入内存。
- 你可以认为CSV文件的第一行是表头,包含了列名。其中,第一列的列名是
name,其余列是不同的STR序列。
- 你可以认为CSV文件的第一行是表头,包含了列名。其中,第一列的列名是
- 你的程序应打开DNA序列并将其内容读入内存。
- 对于CSV文件第一行中的每个STR,你的程序需要计算出在待识别的DNA序列中,该STR连续重复出现的最长次数。注意,我们已经为你定义好了一个辅助函数
longest_match,它可以帮你完成这个计算! - 如果计算出的STR计数与CSV文件中某个人的计数完全一致,程序就应该输出这个人的姓名。
- 你可以假定STR计数与多个个体不匹配。
- 如果STR计数与CSV文件中的任何个体的计数都不完全匹配,则你的程序应打印
No match。
演练
用法
你的程序应按以下示例运行:
$ python dna.py databases/large.csv sequences/5.txt
Lavender
$ python dna.py
Usage: python dna.py data.csv sequence.txt
$ python dna.py data.csv
Usage: python dna.py data.csv sequence.txt
提示
- 你可能会发现Python的
csv模块对于将CSV文件读入内存很有用。你可能需要利用csv.reader或csv.DictReader。 open和read函数可能对于将文本文件读入内存很有用。- 考虑哪些数据结构可能有助于跟踪程序中的信息。
list或dict可能会很有用。 - 请记住,我们定义了一个函数(
longest_match),该函数在给定DNA序列和STR作为输入的情况下,返回STR重复的最大次数。然后,你可以在程序的其他部分使用该函数!
测试
虽然check50可用于此问题,但我们鼓励你首先针对以下各项自行测试代码。
- 运行
python dna.py databases/small.csv sequences/1.txt,程序应输出Bob。 - 运行
python dna.py databases/small.csv sequences/2.txt,程序应输出No match。 - 运行
python dna.py databases/small.csv sequences/3.txt,程序应输出No match。 - 运行
python dna.py databases/small.csv sequences/4.txt,程序应输出Alice。 - 运行
python dna.py databases/large.csv sequences/5.txt,程序应输出Lavender。 - 运行
python dna.py databases/large.csv sequences/6.txt,程序应输出Luna。 - 运行
python dna.py databases/large.csv sequences/7.txt,程序应输出Ron。 - 运行
python dna.py databases/large.csv sequences/8.txt,程序应输出Ginny。 - 运行
python dna.py databases/large.csv sequences/9.txt,程序应输出Draco。 - 运行
python dna.py databases/large.csv sequences/10.txt,程序应输出Albus。 - 运行
python dna.py databases/large.csv sequences/11.txt,程序应输出Hermione。 - 运行
python dna.py databases/large.csv sequences/12.txt,程序应输出Lily。 - 运行
python dna.py databases/large.csv sequences/13.txt,程序应输出No match。 - 运行
python dna.py databases/large.csv sequences/14.txt,程序应输出Severus。 - 运行
python dna.py databases/large.csv sequences/15.txt,程序应输出Sirius。 - 运行
python dna.py databases/large.csv sequences/16.txt,程序应输出No match。 - 运行
python dna.py databases/large.csv sequences/17.txt,程序应输出Harry。 - 运行
python dna.py databases/large.csv sequences/18.txt,程序应输出No match。 - 运行
python dna.py databases/large.csv sequences/19.txt,程序应输出Fred。 - 运行
python dna.py databases/large.csv sequences/20.txt,程序应输出No match。
执行以下命令,使用 check50 评估代码的正确性。 但请务必自行编译和测试!
check50 cs50/problems/2023/x/dna
执行以下命令,使用 style50 评估代码的风格。
如何提交
在您的终端中,执行以下命令以提交您的作品。
submit50 cs50/problems/2023/x/dna